Cluster de calcul (Slurm)
Le cluster de calcul
Nous avons au CEREMADE un cluster pour le calcul parallèle.
Présentation de formation à l’utilisation du cluster
- 09 octobre 2025 : Diapositives cluster.pdf
Description de la structure
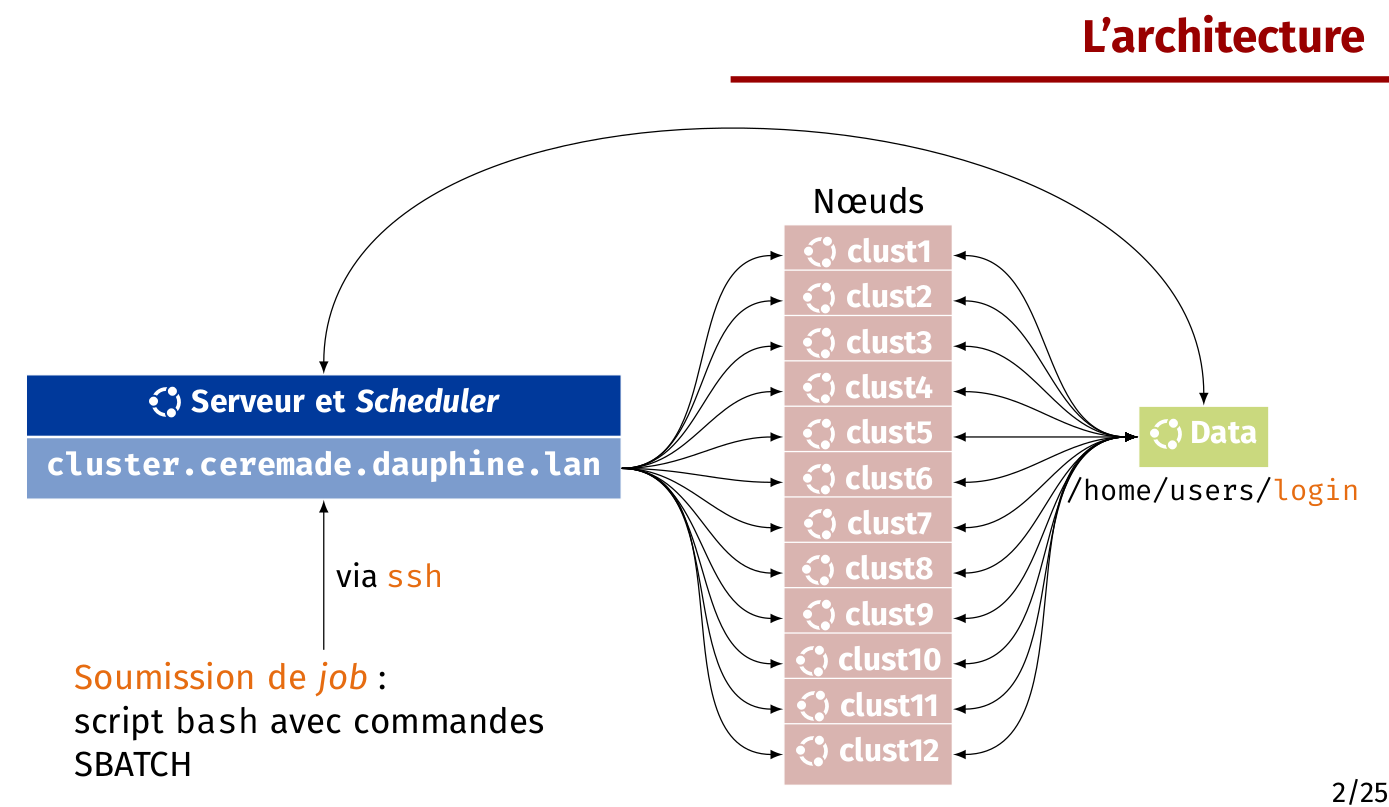
Les nœuds
clust1: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Tesla T4clust2: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Tesla T4clust3: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Tesla P4clust4: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Tesla P4clust5: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Tesla P4clust6: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Tesla P4clust7: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Tesla P4clust8: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Tesla P4clust9: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHzclust10: 20 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHzclust11: 40 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Quadro RTX 4000clust12: 40 cœurs à 2 threads par cœur (40 threads), Intel(R) Xeon(R) CPU E5-2630 v4 @ 2.20GHz, 1 GPU Quadro RTX 4000
Visualiser l’occupation des nœuds
- CPU/RAM : https://www.ceremade.dauphine.fr/affichage/cluster/cluster.html
- GPU/Mémoire : https://www.ceremade.dauphine.fr/affichage/cluster/cluster_gpu.html
SLURM et la machine cluster3.ceremade.dauphine.lan
Pour soumettre un calcul, le service SLURM a été mis en place pour gérer les calculs soumis. Cela s'est fait par la mise en place d'une machine particulière (frontale) nommée cluster3.ceremade.dauphine.lan par laquelle on doit soumettre le calcul désiré en demandant du temps et des ressources qui seront gérés par le service SLURM.
Le cluster de calcul est donc un ensemble de machine dédiées au calcul. Ce n’est pas un ordinateur personnel, et il n’est accessible qu’en ligne de commande. Ainsi, les aspects graphiques de vos recherches ne doivent pas être produits sur ces machines qui n’ont pas d’interface graphique. La production de graphiques doit donc se faire sur vos machines personnelles à partir des données produites lors du calcul sur le cluster.
Copier les fichiers vers cluster3
On peut par exemple envoyer notre répertoire pi contenant le code, les données, etc. par scp:
scp -r /home/chupin/pi/ chupin@cluster3.ceremade.dauphine.lan:~/Il est aussi possible de faire cela via sFTP (en utilisant par exemple FileZilla).
Connexion à la machine cluster3
L'accès au cluster n'est possible que par le biais du VPN
On se connecte à la machine cluster3.ceremade.dauphine.lan par ssh:
ssh nomutilisateur@cluster3.ceremade.dauphine.lansinon
ssh nomutilisateur@10.101.7.24Construction du script SBATCH
Pour envoyer un calcul, il faut écrire un fichier SBATCH qui va indiquer à Slurm les besoins et les commandes pour exécuter le calcul.
Les scripts SBATCH sont des scripts bash qui contiennent des commandes SBATCH sous forme de commentaires.
Exécution du programme de calcul
Pour lancer un job à partir de cluster3.ceremade.dauphine.lan, il faut utiliser sur un fichier dit SBATCH (qui est du bash augmenté) qui pour l'exemple s'appelera submission.SBATCH :
chupin@cluster3:~/pi/> sbatch submission.SBATCHLes autres outils de SLURM pour visualiser, annuler, stopper, etc. un job sont décrits en fin de page.
Exemple minimal de commandes SBATCH
On fournit ici un exemple minimal de commandes SBATCH. Toutes les commandes sont décrite en fin de page.
#!/bin/sh
# Fichier submission.SBATCH
#SBATCH --nodes=1
#SBATCH -c 20
#SBATCH --job-name="MON_JOB"
#SBATCH --output=test.out
#SBATCH --mail-user=chupin@ceremade.dauphine.fr
#SBATCH --mail-type=BEGIN,END,FAILOn demande le nombre de « cœurs » avec l'option -c 20, où on a demandé 20 cœurs ici. En réalité, cette commande permet de choisir le nombre de threads total. Pour les utilisateurs et utilisatrices les plus avancé·e·s, on pourra régler tout ceci très finement avec les options suivantes :
--sockets-per-node=S number of sockets per node to allocate
--cores-per-socket=C number of cores per socket to allocate
--threads-per-core=T number of threads per core to allocateQuelques variables d'environnement de SBATCH
Le scripts SBATCH suivant :
#!/bin/sh
#SBATCH --nodes=1
#SBATCH -c 40
#SBATCH --job-name="Test_Variable"
#SBATCH --output=%x.%J.out
#SBATCH --error=%x.%J.out
#SBATCH --mail-user=chupin@ceremade.dauphine.fr
#SBATCH --mail-type=STAGE_OUT
### Quelques infos qui peuvent etre utiles >
echo Host `hostname`
### Nombre total de CPU
echo Il a ete aloue $SLURM_JOB_CPUS_PER_NODE cpusproduira le résultat suivant :
Host clust3
Il a ete aloue 40 cpusD’autres variables d'environnement sont décrites plus bas.
Exemples complets
Exemple 1 : exemple utilisant OpenMP
Considérons, par exemple, un code C++ compute_pi.cpp utilisant la librairie omp.h et donc les instruction OpenMP au compilateur (mais un code Python rentre aussi dans ce cadre).
Un tel code doit être compilé de la façon suivante :
g++ -o compute_pi -fopenmp compute_pi.cpp Une fois cela fait, on crée un script SBATCH (dans un fichier nommé pour l'exemple submission.SBATCH) qui peut ressembler à :
#!/bin/sh
# fichier submission.SBATCH
#SBATCH --nodes=1
#SBATCH -c 10
#SBATCH --job-name="Test_OpenMP"
#SBATCH --partition=erc
#SBATCH --output=%x.%J.out
#SBATCH --error=%x.%J.out
#SBATCH --mail-user=chupin@ceremade.dauphine.fr
#SBATCH --mail-type=BEGIN,FAIL,END
# on se place dans le répertoire de soumission
cd ${SLURM_SUBMIT_DIR}
# on execute le programme
./compute_piIci, on demande un nœud (OpenMP), 10 cœurs dont chacun n'utilise qu'un thread (alors qu'ils en disposent de deux). Un fois ces fichiers sur la machine cluster dans un répertoire de son home, on soumet le job en utilisant la commande suivante :
chupin@cluster3:~/pi/> sbatch submission.SBATCHExemple 2 : exemple utilisant Python (jupyter)
Si vous n'etes pas dans les locaux de dauphine relié par cable ethernet, vous devez vous connecter au VPN.
Pour utiliser Jupyter nous devons passer par une session interactive, en executant cette commande :
srun --pty -c 10 -N 1 /bin/bashsi tout ce passe bien vous drevriez voir votre prompt passer de:
duleu@cluster3:~/code/test$à
duleu@clust3:~/code/test$Vous pouvez observez que le nom de machine est maintenant clust3 et plus cluster3. Nous pouvons maintenant lancer un notebook jupyter:
jupyter notebook --ip=0.0.0.0Nous obtenons l'adresse a copier coller dans un navigateur:
[I 11:09:58.871 NotebookApp] JupyterLab extension loaded from /home/users/duleu/anaconda3/lib/python3.7/site-packages/jupyterlab
[I 11:09:58.871 NotebookApp] JupyterLab application directory is /home/users/duleu/anaconda3/share/jupyter/lab
[I 11:09:58.876 NotebookApp] Serving notebooks from local directory: /mnt/nfs/rdata02-users/users/duleu/code/test
[I 11:09:58.876 NotebookApp] The Jupyter Notebook is running at:
[I 11:09:58.876 NotebookApp] http://clust3:8888/?token=673591c461f08d5773353be62416ba33a3468551a8926287
[I 11:09:58.876 NotebookApp] or http://127.0.0.1:8888/?token=673591c461f08d5773353be62416ba33a3468551a8926287
[I 11:09:58.876 NotebookApp] Use Control-C to stop this server and shut down all kernels (twice to skip confirmation).
[W 11:09:58.939 NotebookApp] No web browser found: could not locate runnable browser.
[C 11:09:58.939 NotebookApp]
To access the notebook, open this file in a browser:
file:///mnt/nfs/rdata02-users/users/duleu/.local/share/jupyter/runtime/nbserver-1947447-open.html
Or copy and paste one of these URLs:
http://clust3:8888/?token=673591c461f08d5773353be62416ba33a3468551a8926287
or http://127.0.0.1:8888/?token=673591c461f08d5773353be62416ba33a3468551a8926287Il faut rajouter .ceremade.dauphine.lan après clust3.
Exemple 3 : exemple Python
On veut exécuter un programme python script.py sur un nœud et 40 threads. Pour cela, on pourra s'inspirer du fichier SBATCH ci-dessous:
#!/bin/sh
# fichier submission.SBATCH
#SBATCH --nodes=1
#SBATCH -c 20
#SBATCH --job-name="Test_Python"
#SBATCH --output=%x.%J.out
#SBATCH --time=10:00
#SBATCH --error=%x.%J.out
#SBATCH --mail-user=chupin@ceremade.dauphine.fr
#SBATCH --mail-type=BEGIN,FAIL,END
# on se place dans le répertoire de soumission
cd ${SLURM_SUBMIT_DIR}
python3 script.pyExemple 4 : exemple utilisant Matlab
Matlab est installé sur tous les nœuds du cluster3. On peut donc l'utiliser. Supposons que dans notre répertoire de travail, nous ayons un script script.m que l'on veut exécuter.
#!/bin/sh
#SBATCH --nodes=1
#SBATCH -c 20
#SBATCH --job-name="Test_Matlab"
#SBATCH --output=%x.%J.out
#SBATCH --error=%x.%J.out
#SBATCH --mail-user=chupin@ceremade.dauphine.fr
#SBATCH --mail-type=BEGIN,FAIL,END
# on se place dans le répertoire de soumission
cd ${SLURM_SUBMIT_DIR}
matlab -nodisplay -nodesktop -r "run('test.m')"Attention : ici, nous avons demandé 1 nœud avec 20 cœurs (--ntasks-per-node=20) mais un seul thread par cœur (--cpus-per-task=1) en effait pour pouvoir utiliser les 40 threads disponibles, il faut faire du multithreading avec Matlab, et nous ne savons pas faire sans interface graphique.
Attention : sur quelques comptes, matlab n'est pas accessible, et il faut spécifier le chemin complet de l'exécutable :
/usr/local/bin/matlab -nodisplay -nodesktop -r "run('script.m')"Exemple 5 : exemple utilisant un GPU (carte graphique)
Pour demander des ressource GPU il est necessaire de rajouter cette information dans le fichier pbs ou lors d'une session interactive.
voici un exemple de fichier avec demande de ressource GPU:
#!/bin/bash >
# Fichier submission.SBATCH
#SBATCH --nodes=1
#SBATCH -c 20
#SBATCH --gres=gpu:1
#SBATCH --job-name="MON_JOB"
#SBATCH --output=%x.%J.out
#SBATCH --error=%x.%J.out
#SBATCH --mail-user=<nom>@ceremade.dauphine.fr
#SBATCH --mail-type=BEGIN,END,FAIL
# on se place dans le répertoire de soumission
cd ${SLURM_SUBMIT_DIR}
# Pour l'export OpenMP
OMP_NUM_THREADS=$SLURM_JOB_CPUS_PER_NODE
export OMP_NUM_THREADS
# on se deplace dans le repertoire de SLURM
cd $SLURM_SUBMIT_DIR
# on execute le programme matlab mais sans interface graphique
matlab -nodisplay -nodesktop -r "run('script.m')"
l'instruction nécessaire est la suivante : --gres=gpu:1.
Exemple 6 : avec R
#!/bin/bash
#SBATCH --job-name "demo_r"
#SBATCH --cpus-per-task=4
#SBATCH --mail-user="nom@ceremade.dauphine.fr"
#SBATCH --ntasks=1
#SBATCH --nodes=1
#SBATCH --mail-type=BEGIN,END,FAIL
#SBATCH --partition=debug
# on se place dans le répertoire de soumission
cd ${SLURM_SUBMIT_DIR}
Rscript demo_r2.RConnection en mode interactif
Pour ouvrir une session interactive il faut utiliser la commande srun avec les options décrites plus bas. Par exemple :
srun --pty -c 10 -N 1 /bin/bashSi tout ce passe bien vous drevriez voir votre prompt passer de:
duleu@cluster3:~/code/test$à
duleu@clust3:~/code/test$Compilation des langages compilés
La machine frontale cluster3.ceremade.dauphine.lan n'est pas adaptée pour compiler les programmes. On utilisera ce mode interactif pour compiler les sources dans les langages compilés (tels que C, C++, Fortran, etc.). Cela permettra notamment d’obtenir des binaires cohérents avec l’architecture de calcul.
srun --pty -c 10 -N 1 /bin/bashpuis pour un code en C++ :
utilisateur@clust3:~/code/test$ g++ -o compute_pi -fopenmp compute_pi.cpp Les packages et autres bibliothèques des langages non compilés
Si vous utilisez Python, Julia, R, et que votre code utilise des bibliothèques particulières, elle s’installent localement dans votre home (avec pip, Pkg, etc.).
Pour ce faire, il faut donc utiliser un srun pour avoir accès à un nœud (n’importe lequel, le home est partagé sur l’ensemble des nœuds), puis exécuter vos commandes usuelles pour l’installation de vos bibliothèques préférées.
Les options de SBATCH
Pour plus d’information sur "sbatch", voir la documentation officielle à l’adresse : https://slurm.schedmd.com/sbatch.html
| Option | Raccourcis | Description |
|---|---|---|
#SBATCH --partition=<part> |
-p <partition> |
Choix de la partition à utiliser pour le job |
#SBATCH --job-name=<name> |
-J <name> |
Définit le nom du job tel qu'il sera affiché dans les différentes commandes Slurm (squeue, sstat, sacct) |
#SBATCH --output=<stdOutFile> |
La sortie standard (stdOut) sera redirigée vers le fichier défini par "--output" ou, si non définie, un fichier par défaut "slurm-%j.out" (Slurm remplacera "%j" par le JobID). | |
#SBATCH --error=<stdErrFile> |
La sortie d'erreur (stdErr) sera redirigée vers le fichier défini par "--error" ou, si non définie, vers la sortie standard. | |
#SBATCH --input=<stdInFile> |
L'entrée standard peut aussi être redirigée avec "--input". Par défaut, "/dev/null" est utilisé (aucune/vide). | |
#SBATCH --open-mode=<append,truncate> |
L'option "--open-mode" définit le mode d'ouverture (écriture) des fichiers et se comporte comme un open/fopen de la plupart des langages de programmation (2 possibilités : "append" pour écrire à la suite du fichier (s'il existe) et "truncate" pour écraser le fichier à chaque exécution du batch (valeur par défaut)). | |
#SBATCH --mail-user=<e-mail> |
||
#SBATCH --mail-type=<BEGIN,END,FAIL,TIME_LIMIT,TIME_LIMIT_50,...> |
Permet d'être notifié par e-mail d'un évènement particulier dans la vie du job : début de l'exécution (BEGIN), fin d'exécution (END, FAIL et TIME_LIMIT)... Consulter la documentation Slurm pour la liste complète des évènements pris en charge. | |
--sockets-per-node=<n> |
Nombre de sockets par node | |
--cores-per-socket=<n> |
Nombre de cœurs par sockets | |
--threads-per-core=<n> |
Nombre de threads par cœur | |
#SBATCH --cpus-per-task=<n> |
-c <n> |
Définit le nombre de CPUs à allouer par Task. L'utilisation effective de ces CPUs est à la charge de chaque Task (création de processus et/ou threads). |
#SBATCH --ntasks=<n> |
-n <n> |
Definit le nombre maximum de Tasks exécutées en parallèle. |
#SBATCH --mem-per-cpu=<n> |
Définit la RAM en Mo allouée à chaque CPU. Par défaut, 4096 Mo sont alloués à chaque CPU, l’utilisation de cette variable permet de spécifier une taille de RAM spécifique, inférieure ou égale à 7800 Mo (maximum allouable par CPU) | |
#SBATCH --nodes=<minnodes[-maxnodes]> |
-N <n> |
Nombre minimum[-maximum] de nœuds sur lesquels distribuer les Tasks. |
#SBATCH --ntasks-per-node=<n> |
Utilisée conjointement avec --nodes, cette option est une alternative à --ntasks qui permet de contrôler la distribution des Tasks sur les différents noeuds. |
Les variables d'environnement de SBATCH
| Nom de la variable | Description | Exemple |
|---|---|---|
SLURM_JOB_ID |
L'identifiant du job (calcul) | 12345.cluster |
SLURM_JOB_NAME |
Nom du job défini avec l'option -J |
my_job |
SLURM_JOB_NODELIST |
Nom d'un fichier qui est fabriqué par SLURM et qui contient la liste des nœuds utilisés. | |
SLURM_SUBMIT_HOST |
Nom de l'hôte sur lequel sbatch a été exécuté (chez nous cluster) |
cluster |
SLURM_SUBMIT_DIR |
Répertoire depuis lequel le job est soumis | /home/user/chupin/scripts_pbs |
SLURM_JOB_NUM_NODES |
Nombre de nœuds requis pour le job (par exemple avec -N 5) |
|
SLURM_NTASKS_PER_NODE |
Nombre de cœurs par nœud requis pour le job | |
SLURM_JOB_CPUS_PER_NODE |
Nombre total de threads par nœud |
Visualiser les jobs
Pour lister les calculs lancés sur le cluster, on utilise le programme ctop :
chupin@cluster3:~/pi/> ctop -t -s 1qui produit quelque chose comme :
Usage Totals: 314/560 Cores | 7/12 Nodes | 16/16 Jobs Running 2023-06-29-T15:52:17
Node States: 1 ALLOCATED | 2 DOWN* | 3 IDLE | 6 MIXED
cluster node 1 2 3 4 5 6 7 8 9 0 1 2 3 4 5 6 7 8 9 0 1 2 3 4 5 6 7 8 9 0 1 2 3 4 5 6 7 8 9 0 load
-------------------------------------------------------------------------------------
cluster clust1 B B B B B B B B B B B B B B B B B B B B B B B B B B M M M M M M M M @ @ @ @ @ @ 5.58 = too low!
-------------------------------------------------------------------------------------
cluster clust10 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 0.25 = Ok
-------------------------------------------------------------------------------------
cluster clust11 r r S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S S
cluster clust11 S S H H H H H H H H H H H H H H H H H H H H H H H H H H @ @ @ @ @ @ @ @ @ @ @ @ 41.21 = too low!
-------------------------------------------------------------------------------------
cluster clust12 A A A A A A A A A A A A A A A A A A A A A A A A A A t t t t t t t t t t t t t t
cluster clust12 t t t t t t t t t t t t t t t t t t t t t t t t t t @ @ @ @ @ @ @ @ @ @ @ @ @ @ 1.06 = too low!
-------------------------------------------------------------------------------------
cluster clust2 I I I I I I I I I I I I I I I I I I I I T T T T T T T T T T T T T T T T T T T T 80.96 = TOO HIGH!
-------------------------------------------------------------------------------------
cluster clust3 . . . . @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ @ 1.09 = Ok
-------------------------------------------------------------------------------------
cluster clust4 Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z Z D D D D D D D D @ @ @ @ @ @ 7.49 = too low!
-------------------------------------------------------------------------------------
cluster clust5 z z z z z z z z z z z z z z z z z z z z z z z z z z m m m m m m m m s s s s @ @ 5.00 = too low!
-------------------------------------------------------------------------------------
cluster clust6 E E E E E E E E E E E E E E E E E E E E E E E E E E C C C C C C C C @ @ @ @ @ @ 3.56 = too low!
-------------------------------------------------------------------------------------
cluster clust7 X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X N/A = ?
-------------------------------------------------------------------------------------
cluster clust8 X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X X N/A = ?
-------------------------------------------------------------------------------------
cluster clust9 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 0.25 = Ok
-------------------------------------------------------------------------------------
legend: ? unknown | @ busy | X down | . idle | 0 offline | ! other
JobID Username QoS Jobname S Walltime Used
t = 5 turinici normal jng4 R 9-19:21:27
r = 16 ruan normal bash R 8-04:52:16
s = 48 susmann normal qsl R 6-22:24:07
S = 175 susmann normal qslsimol R 3-01:09:32
I = 197 turinici normal LL_1Dpl_s10_p12 R 2-00:12:25
T = 199 turinici normal LL_1Dpl_s10_p14 R 2-00:11:31
H = 202 zarhali normal GMM calib Q15_8_ qvMRMgij0.01 calibratio R 2-00:08:11
A = 203 zarhali normal GMM calib Q15_8_ qvMRWgij0.01 calibratio R 1-23:59:10
E = 207 zarhali normal GMM calib Q25_8_ qvMRMgij0.1 new taulist R 1-21:05:36
z = 208 zarhali normal GMM calib Q25_8_ nonqvgij0.1 new taulist R 1-17:44:15
B = 209 zarhali normal GMM calib Q25_8_ qvMRWgij0.1 new taulist R 0-10:04:51
Z = 211 zarhali normal GMM calib Q25_8_ nonqvgij0.4 new taulist R 0-09:02:51
M = 225 makaroff normal simvae_mean R 0-05:58:34
D = 226 makaroff normal simvae_mean R 0-05:42:04
m = 227 makaroff normal simvae_mean R 0-05:26:34
C = 229 makaroff normal simvae_mean R 0-02:26:46Les options pour ctop sont les suivantes :
Options:
-u [user] Show only jobs for a subset of users.
Comma separated list.
-n [node] Show only jobs for a subset of nodes.
Comma separated list.
-c [type] Color cores in grid display by type.
Type must be one of 'job', 'node', 'qos' or resources.
-C Toggle colorization.
-S Toggle state summary display.
-G Toggle grid display.
-Q Toggle queue display.
-t Toggle showing queued jobs in queue display.
-j Toggle jobs in grid display.
-s [0-9]+ Seconds between refreshes.
-w [0-9]+ With of grid in number of cores to display.
-V Print version and exit.
-h Print this help and exit.Pour voir le taux d'occupation des noeuds on utilise la commande pestat :
chupin@cluster3:~/pi/> pestatVoici un exemple de ce qui s'affiche:
Hostname Partition Node Num_CPU CPUload Memsize Freemem Joblist
State Use/Tot (MB) (MB) JobId User ...
clust1 debug* down* 0 40 0.00* 1 0
clust2 debug* down* 0 40 0.00* 1 0
clust3 debug* idle 0 40 0.20 1 87878
clust4 debug* down* 0 40 0.00* 1 0
clust5 debug* down* 0 40 0.00* 1 0
clust6 debug* down* 0 40 0.00* 1 0
clust7 debug* down* 0 40 0.00* 1 0
clust8 debug* down* 0 40 0.00* 1 0
clust9 erc down* 0 40 0.00* 1 0
clust10 erc down* 0 40 0.00* 1 0 Les programmes de gestion de jobs
SLURM fournit plusieurs programmes pour gérer les jobs.
- sbatch qui permet de soumettre un job.
- srun qui permet de lancer un job en direct.
- sstat qui examine le statut d'un job. Il faut donné l'ID donné dans la colonne
#JOBIDdesmap.chupin@cluster3:~/pi/> sstat 150 - scancel qui supprime un job. Il faut donné l'ID donné dans la colonne
#JOBIDdesmap.